Kopieren leicht gemacht: Eine universelle isotherme Methode zur Amplifikation von DNA
Ob ein Täter anhand von DNA-Spuren entlarvt oder ein Krankheitserreger diagnostiziert, ob ein paläontologischer Fund zugeordnet oder eine Vaterschaft geklärt werden soll – eine Vervielfältigung von Nukleinsäuren (Amplifizierung) ist unabdingbar. In der Zeitschrift Angewandte Chemie stellen Wissenschaftler jetzt eine neue, sehr einfache, aber empfindliche und zuverlässige Methode vor, die ohne die sonst üblichen Heiz- und Kühlschritte oder komplizierte Geräte auskommt. Reagenzien können gefriergetrocknet werden, sodass die universelle Methode problemlos auch außerhalb des Labors verwendet werden kann.
© Wiley-VCH
Die am weitesten verbreitete Amplifikationsmethode, die Polymerase-Kettenreaktion (polymerase chain reaction, PCR), basiert auf dem Durchlaufen wiederholter Temperaturzyklen in speziellen Geräten mit hohem Strombedarf. Außerhalb des Labors, etwa direkt am Krankenbett oder an abgelegenen Orten, ist eine Durchführung schwierig. Alternative Methoden ohne Temperaturzyklen sind dafür oft kompliziert oder wenig empfindlich, benötigen teure Reagenzien oder sind nicht breit anwendbar.
Die Forscher um Bin-Cheng Yin und Bang-Ce Ye von der East China University of Science & Technology in Shanghai haben jetzt eine neue, kostengünstige Methode entwickelt, Cas9n-based amplification reaction (Cas9nAR) genannt, die nur aus einem Schritt in einer homogenen Lösung besteht und bei konstanten 37 °C abläuft.
Die Forscher verwenden für ihren Ansatz Bestandteile des „Immunsystems“ von Bakterien. Werden Bakterien z.B. von Viren infiziert, spalten sie das fremde Erbgut in kleine Schnipsel und fügen diese an bestimmten Stellen in das eigene Genom ein. Bei einem erneuten Befall „erkennen“ RNA-Stränge des Bakteriums diese Sequenzen wieder und steuern spezielle „Genscheren“, um die Fremd-DNA zu zerschneiden. Inzwischen nutzt man diese Werkzeuge auch in der Gentechnik.
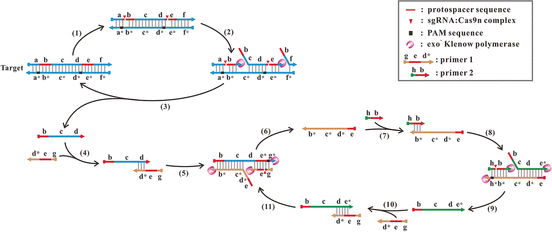
Yin, Ye und Kollegen haben die bakterielle Genschere Cas9 so verändert, dass sie die DNA nicht mehr komplett durchschneidet, sondern nur einen Strang, die DNA quasi „einkerbt“. Ein solcher Enzymtyp wird als „Nickase“ bezeichnet (engl. nick: Kerbe). Wie beim bakteriellen Immunsystem auch bindet die Cas9-Nickase einen RNA-Strang, der die Kerbestelle bestimmt. Diese RNA kann nun so konstruiert werden, dass sie eine z.B. für ein Pathogen charakteristische DNA-Sequenz erkennt. Die Cas9-Nickase kerbt die DNA dann in direkter Nachbarschaft ein.
Für die neue Methode werden zwei verschiedene Cas9-Nickase-RNA-Komplexe hergestellt, die die DNA an zwei verschiedenen Stellen einkerben. Eine in der PCR gängige Polymerase (exo−-Klenow-Polymerase) vervollständigt den zerschnittenen Strang dann ausgehend von der ersten Kerbe und löst dabei den alten Strang Stück für Stück ab, bis sie an der zweiten Kerbe angelangt ist. Die so vervollständigte DNA wird nun immer wieder erneut vom Nickase-Komplex eingekerbt und ergänzt. Die auf diese Weise freigesetzten kurzen Einzelstränge werden in einem zweiten Zyklus zum Ausgangspukt für eine weitere Vervielfältigung. Zusätzlich zum Nickase-Komplex und der Polymerase werden nur zwei passende Primer als Startpunkte für die Kopien benötigt.
Tests mit einem Fragment einer bakteriellen genomischen DNA zeigten, dass die Zielsequenz präzise erkannt und amplifiziert wird. In einem 20 μl-Volumen ließ sich so eine Einzelmolekül-Detektion erreichen. Unterschiede eines einzelnen Nukleotids innerhalb eines Gens ließen sich hochspezifisch nachweisen.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.