Proteinen bei der Arbeit zugesehen: Calmodulin, ein Protein packt zu
Calmodulin ist der klangvolle Name eines der häufigsten Proteine in unseren Zellen. Es steuert die Aktivität vieler Hundert anderer Eiweiße. Mit einer an der Technischen Universität München (TUM) entwickelten Methode ist es nun erstmals möglich, dem Protein bei seiner Arbeit zuzuschauen. Mit einem speziellen Kraft-Mikroskop können Forscher der TU München direkt die mechanischen Veränderungen eines einzelnen Calmodulin-Moleküls bei der Anlagerung an andere Eiweiße Messen.
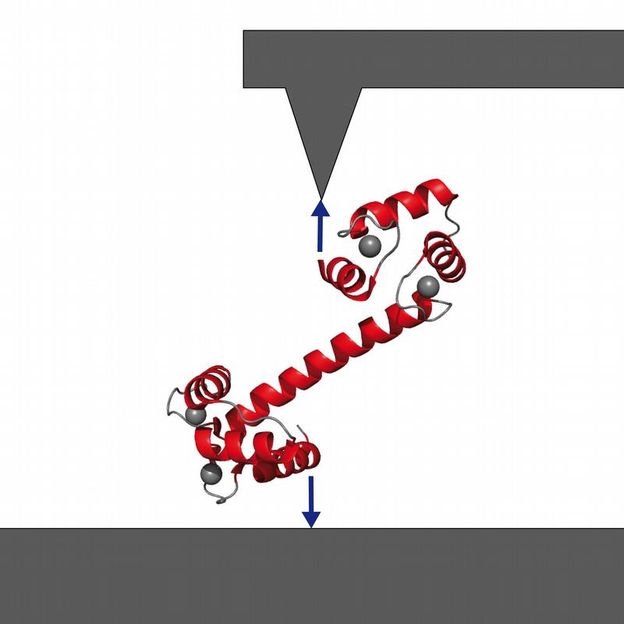
Den Wissenschaftlern ist es gelungen, ein einzelnes Calmodulin an der Spitze eines Kraft-Mikroskops zu befestigen. Die mechanischen Eigenschaften des Proteins, die sie so unter verschiedensten Bedingungen messen können, geben tiefe Einblicke in die Arbeitsweise des Proteins.
TUM/Rief/Junker
Seit langem gilt das Eiweiß Calmodulin als eines der wichtigsten Signalmoleküle. Indem es sich anlagert oder wieder ablöst gibt es Start- und Stop-Signale für eine große Zahl von Proteinaktivitäten. Calmodulin kann bis zu vier Calcium-Ionen binden. Der Körper nutzt dies, um die Aktivität des Calmodulins zu kontrollieren. Je nach dem, wie viele Calcium-Ionen an das Calmodulin gebunden sind, nimmt es eine unterschiedliche räumliche Struktur ein. Und je nach seiner Struktur kann es sich an unterschiedliche Aminosäureketten von Proteinen anlagern.
Die Garchinger Forscher haben es nun geschafft, ein einzelnes Calmodulin-Molekül auf der einen Seite auf einer Platte zu fixieren und auf der anderen Seite an die Spitze eines Kraft-Mikroskops anzuhängen. Mit dieser Anordnung können Sie nun dem Molekül bei der Arbeit zuschauen. Geben die Wissenschaftler Calcium-Ionen in die Lösung, so dehnt sich das Calmodulin aus und bindet vier Calcium-Ionen. Es hat nun völlig andere mechanische Eigenschaften, die die Forscher mit ihrem Mikroskop gut verfolgen können. Auch die Bindung an Aminosäureketten testeten die Forscher bereits und konnten damit zeigen, wie das Calmodulin bestimmte Ketten erkennt und sich an diese anlagert.
"Das Besondere an unserer Technologie ist, dass wir direkt in wässriger Lösung arbeiten können," sagt Professor Matthias Rief, der die Arbeitsgruppe leitet. "Wir können genau unter den Bedingungen messen, unter denen das Protein auch in seiner natürlichen Umgebung arbeitet." Die Methode ist damit eine wertvolle Ergänzung der Röntgenstrukturanalyse, die zwar die genaue räumliche Struktur eines Proteins zeigt, aber dafür geordnete Kristalle braucht. Von dynamischen Vorgängen kann Röntgenstrukturanalyse allenfalls Momentaufnahmen liefern. "Wir können nun direkt beobachten, wie das Calmodulin sich die Aminosäurekette schnappt und sich umfaltet, um diese festzuhalten."
Mit ihrem Kraft-Mikroskop können die Wissenschaftler um Matthias Rief direkt die Kräfte messen, die man benötigt, um das Calmodulin aus der jeweils stabilen Lage heraus umzufalten. Daraus können sie die Energien berechnen, die bei der Anlagerung von Calcium und Aminosäureketten an das Calmodulin auftreten. Und indem sie die mechanischen Eigenschaften über eine gewisse Zeit verfolgen, können sie auch feststellen, wie lange ein anderes Proteinfragment gebunden bleibt.
"Wir haben mit der Entwicklung dieser Methode ein Fenster aufgestoßen, durch das wir sehr viel über die grundsätzlichen Abläufe in der Zelle erfahren können," sagt Rief. "Es ist unglaublich faszinierend, direkt die dynamischen Abläufe ansehen zu können. Das Calmodulin wird nicht das einzige Molekül bleiben, das wir uns genauer ansehen."
Originalveröffentlichung: Jan Philipp Junker, Fabian Ziegler, Matthias Rief; "Ligand-Dependent Equilibrium Fluctuations of Single Calmodulin Molecules" Science, 30 January 2009, Vol. 323. no. 5914, pp. 593 - 594
Meistgelesene News
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.


















































