Jedes Atom zählt: Forscher machen Proteasom in zuvor unerreichter Detailschärfe in 3D sichtbar
Detaillierte 3D-Struktur des zellulären Abfallverwerters enthüllt, wie dieser durch Krebsmedikamente blockiert wird
Bösartige Krebszellen wachsen schneller als die meisten unserer Körperzellen. Sie produzieren deshalb auch mehr „Abfall“ wie etwa ausgediente Proteine. Dies macht Krebszellen besonders abhängig von dem wichtigsten zellulären Müllverwerter, dem Proteasom, das fehlerhafte und nicht mehr benötigte Proteine in ihre Bausteine zerlegt. Bei der Behandlung mancher Krebsarten, etwa dem Multiplen Myelom – einem Tumor des Knochenmarks – macht man sich das zunutze: Patienten werden unter anderem mit sogenannten Inhibitoren behandelt, die das Proteasom gezielt blockieren. Der folgende Entsorgungsstau stürzt die Krebszelle ins Chaos und führt schließlich dazu, dass sie stirbt. Ein Forscherteam aus Göttingen und Hamburg hat das Proteasom jetzt in zuvor unerreichter Detailschärfe in 3D sichtbar gemacht und den genauen Mechanismus entschlüsselt, mit dem Inhibitoren das Proteasom hemmen. Die neuen Erkenntnisse sind wegweisend, um effektivere Proteasom-Inhibitoren für die Krebstherapie entwickeln zu können.
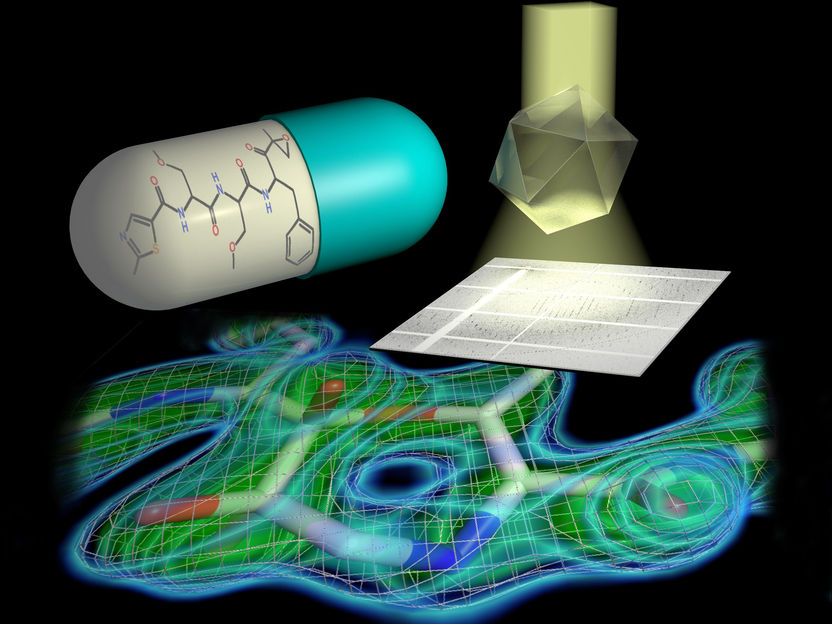
Parallele Röntgenstrahlen, die maßgeschneidert auf die Abmessung der Proteinkristalle passen, ermöglichten es den Wissenschaftlern, die Struktur des Proteasoms in bisher unerreichtem Detail aufzuklären (oben rechts). Sie konnten nachweisen, dass bei der chemischen Reaktion von Inhibitor und Proteasom ein 7-Ring entsteht (unten). Diese Erkenntnis ermöglicht das Design neuer Therapeutika (oben links).
© Hartmut Sebesse / Max Planck Institute for Biophysical Chemistry
Wie genau zelluläre Maschinen wie das Proteasom funktionieren, lässt sich nur verstehen, wenn man ihren räumlichen Aufbau im Detail kennt. Mit seinen mehr als 50.000 Atomen ist der tonnenförmige Müllverwerter für Strukturbiologen allerdings eine echte Herausforderung. Wissenschaftlern um Ashwin Chari vom Göttinger Max-Planck-Institut (MPI) für biophysikalische Chemie und Gleb Bourenkov vom European Molecular Biology Laboratory (EMBL) ist es nun mittels Röntgenkristallografie gelungen, die dreidimensionale Struktur des menschlichen Proteasoms mit einer Trennschärfe von bis zu 1,8 Ångström aufzuklären – und damit die einzelnen Atome des Müllverwerters sichtbar zu machen.
Im nächsten Schritt bestimmten die Forscher außerdem die Struktur des Proteasoms gebunden von vier verschiedenen Inhibitoren, die bereits klinisch im Einsatz sind oder derzeit in Studien getestet werden. „Dank der stark verbesserten Auflösung im Vergleich zu früheren Proteasom-Strukturen konnten wir erstmals den genauen chemischen Mechanismus ermitteln, mit dem die Inhibitoren das Proteasom blockieren. Dieses Wissen ermöglicht es, das Design der Inhibitoren und damit deren Wirksamkeit zu optimieren. Denn nur maßgeschneiderte Inhibitoren hemmen die Aktivität des Proteasoms perfekt und können es komplett stilllegen“, erklärt Chari, Projektgruppenleiter in der Abteilung Strukturelle Dynamik von Holger Stark am MPI für biophysikalische Chemie.
Ein wichtiges Detail entdeckten die Wissenschaftler im sogenannten aktiven Zentrum des Proteasoms, an dem der zelluläre Müll abgebaut wird und an dem sich auch die Inhibitoren anlagern: Anders als bisher gedacht, entsteht bei der chemischen Reaktion von Inhibitor und Proteasom eine 7-Ring Struktur, die eine zusätzliche sogenannte Methylengruppe enthält – mit weitreichenden Folgen für die Wirksamkeit und den chemischen Mechanismus des Inhibitors, so die Forscher. „Auch wenn es sich bei der Methylengruppe um nur ein Kohlenstoffatom samt zweier benachbarter Protonen unter mehr als 50.000 Atomen im Proteasom handelt, beeinflusst diese ganz wesentlich, wie der Inhibitor chemisch beschaffen sein muss, um das Proteasom optimal zu blockieren“, sagt Thomas Schneider, Gruppenleiter am EMBL. „Das muss man bei der Entwicklung neuer Inhibitoren berücksichtigen und die Suche nach Wirkstoff-Kandidaten entsprechend anpassen“, ergänzt Holger Stark. Das chemische Verfahren, mit dem sich Inhibitoren entsprechend designen lassen, haben die Forscher bereits zum Patent angemeldet. „Da einer möglichen medizinischen Anwendung immer das Erkennen vorausgeht, sind es solche Details, bei denen jedes Atom zählt, die den Unterschied ausmachen“, wie Bourenkov erklärt.
Großer Aufwand zeigt den kleinen Unterschied
Der Erfolg des Projekts ist das Ergebnis großartiger Teamarbeit, betont Max-Planck-Forscher Chari: „Mehrere Wissenschaftler, alle Experten auf ihrem Gebiet, haben ihr jeweiliges Fachwissen beigetragen und sich perfekt ergänzt.“ So arbeiteten für das Projekt Strukturbiologen, Physiker, Kinetiker und Biochemiker des MPI für biophysikalische Chemie, des EMBL in Hamburg und der Universität Göttingen zusammen und entwickelten verschiedene innovative Verfahren.
Um die Struktur eines Moleküls mithilfe von Röntgenkristallografie zu bestimmen, züchten Wissenschaftler von diesem Molekül Kristalle, die sie dann mit Röntgenlicht bestrahlen. Die Röntgenstrahlen werden am Kristall gebeugt und erzeugen ein charakteristisches Muster, anhand dessen sich schließlich die Struktur des Moleküls bestimmen lässt. Doch in der Praxis ist dies weit schwieriger als es klingt. Mithilfe der neuen Methoden gelang es Fabian Henneberg und Jil Schrader, Nachwuchswissenschaftler in Starks Abteilung und Erstautoren der jetzt in Science erschienen Arbeit, die Proteasomen äußerst rein herzustellen und daraus hochqualitative Kristalle des Komplexes mit und ohne gebundenem Inhibitor zu züchten.
Die besondere Reinheit der Proben und die Qualität der Kristalle waren eine entscheidende Voraussetzung, die räumliche Struktur des Müllverwerters derartig detailliert aufklären zu können. Auch das Verfahren zur Aufreinigung und Kristallisation meldeten die Wissenschaftler bereits zum Patent an. „Die Methode, mit der wir das Proteasom aufreinigen und mit und ohne Inhibitor kristallisieren, ist außerdem einsetzbar, um neue Wirkstoffe auf ihre Eignung als Proteasom-Inhibitoren zu testen – im industriellen Maßstab möglicherweise Hunderte pro Woche“, wirft Chari einen Blick in die Zukunft.
Eine zweite entscheidende Voraussetzung für den Erfolg des Projekts war die Brillianz des Röntgenlichts. Dieses lieferte die EMBL-Forschungsanlage am DESY: „Die DESY-Strahlenquelle generiert Röntgenstrahlen von herausragender Qualität. Mithilfe von Hochleistungs-Röntgenoptiken konnten wir die Röntgenstrahlen für das kristallisierte Proteasom maßschneidern und diese hohe Detailschärfe erreichen“, sagt Bourenkov.