SMART: Gesichtserkennung für molekulare Strukturen
Paradigmenwechsel in der Auswertung von NMR-Spektren
Ein interdisziplinäres Forscherteam der University of California San Diego hat eine Methode entwickelt, mit der sich die molekularen Strukturen von Naturprodukten schneller und genauer als mit bisherigen Methoden identifizieren lassen. Die Methode funktioniert wie eine Gesichtserkennung für molekulare Strukturen - sie verwendet ein Spektrum, das für jedes Molekül einzigartig ist, und leitet es dann durch ein tiefliegendes neuronales Netzwerk, um das unbekannte Molekül in einem Cluster von Molekülen mit ähnlichen Strukturen zu platzieren.
Das zum Patent angemeldete neue System trägt den Namen "SMART", was für Small Molecule Accurate Recognition Technology steht und das Potenzial hat, den Prozess der Identifizierung der molekularen Struktur um das Zehnfache zu beschleunigen. Diese Entwicklung könnte einen Paradigmenwechsel in den Bereichen der chemischen Analytik, Pharmazie und Wirkstoffforschung bedeuten, da 70 Prozent aller von der FDA zugelassenen Arzneimittel auf Naturprodukten wie Bodenmikroorganismen, Landpflanzen und zunehmend auch marinen Lebewesen wie Algen basieren.
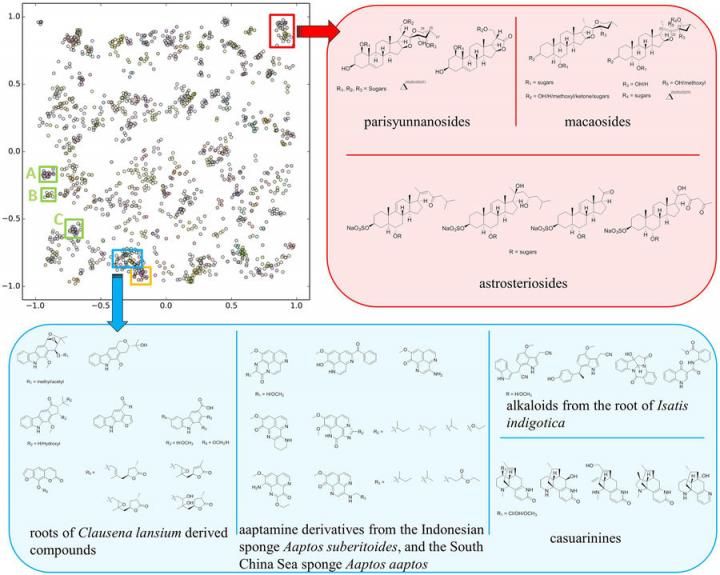
Die SMART-Cluster-Map basiert auf Trainingsergebnissen von 2.054 HSQC-Spektren mit über 83.000 Iterationen, wobei die Inset-Boxen verschiedene Klassen repräsentieren.
Chen Zhang, Bill Gerwick
Diese Arbeit stellt eine Zusammenarbeit zwischen der UC San Diego Jacobs School of Engineering und der UC San Diego Scripps Institution of Oceanography dar.
"Die Struktur eines Moleküls ist die Grundlage für Informationen", sagt Bill Gerwick, Professor für Ozeanographie und Pharmazeutische Wissenschaften an der UC San Diego's Scripps Institution of Oceanography. "Sie müssen die Struktur für jede FDA-Zulassung vorweisen können." Wenn man geistiges Eigentum erwerben will, muss man diese Struktur patentieren lassen, wenn man Analoga aus diesem Molekül machen will, muss man wissen, was das Ausgangsmolekül ist - es ist eine wichtige Information."
Chen Zhang ist Doktorand an der UC San Diego Jacobs School of Engineering. Er erklärt, dass die Bestimmung der Struktur eines Moleküls ein Flaschenhals in der Naturstoffforschung sein kann. Experten brauchen Monate und sogar Jahre, um die richtige und vollständige Struktur genau zu bestimmen. Während jedes Molekül und sein Identifikationszeitraum unterschiedlich sind, gibt der SMART-Ansatz den Forschern einen ersten Anhaltspunkt dafür, unter welche Familie ein neues Molekül fällt.
"Wir konnten den Prozess beschleunigen, indem wir im Wesentlichen Gesichtserkennungssoftware verwenden, um die wichtigsten Informationen über die Moleküle zu erhalten", erklärt Gerwick. Die Schlüsselinformation, die das Team verwendet, ist ein HSQC-Spektrum. Es produziert eine topologische Karte von Spots, die zeigt, welche Protonen im Molekül direkt an welche Kohlenstoffatome gebunden sind und für jedes Molekül einzigartig sind.
Zhang und Gerwick haben sich mit Gary Cottrell, einem Informatik- und Ingenieurprofessor an der UC San Diego Jacobs School of Engineering, zusammengetan, um ein tieflernes System zu entwickeln, das mit Tausenden von HSQC-Spektren aus der Literatur trainiert wurde. Dieses neuronale Konvolutionsnetzwerk nimmt ein 2D-Bild des HSQC-NMR-Spektrums eines unbekannten Moleküls auf und bildet es in einem 10-dimensionalen Raum in der Nähe ähnlicher Moleküle ab, was es den Forschern erleichtert, die Struktur eines unbekannten Moleküls zu verstehen.
"Chen nahm diese Annäherung, um NMR Spektren von über 4.000 Verbindungen von der Literatur zu erhalten, indem er buchstäblich heraus die Bilder aus den Publikationen schnitt," Cottrell sagte. "Es war eine gewaltige Anstrengung! Trotzdem reicht das normalerweise nicht aus, um ein tiefliegendes Netzwerk zu trainieren, aber wir haben eine Technologie namens Siamesisches Netzwerk verwendet, in dem Sie mit Bildpaaren trainieren. Das verstärkt Ihre Schulung um das Quadrat der Anzahl der Verbindungen in einer Familie und macht dieses Projekt erst möglich."
Gerwick hat damit zum ersten Mal einen Ingenieurstudenten als Mentor begleitet und der Austausch erwies sich als fruchtbar.
"Es war eine wundervolle Interaktion. UC San Diego hat etwas wirklich ziemlich magisches in sich. Und das ist die Tiefe der Zusammenarbeit, die zwischen den Abteilungen stattfindet - das ist phänomenal", sagte Gerwick. "Wenn man versucht, aus einer anderen Disziplin etwas zu übernehmen, was in dieser Disziplin vielleicht sogar alltäglich ist, und es auf eine neue und einzigartige Weise in unserer Disziplin anwendet, ist es eine Gelegenheit, diese Art von Paradigmenwechsel-Ding wirklich zu haben. Und ich denke, dass diese Technologie mit einigen Fortschritten ein echter Paradigmenwechsel in der Art und Weise sein könnte, wie wir alle Arten von Chemie und chemischer Analyse durchführen."
Das Team erhält diese Chance auf Weiterentwicklung dank eines Zuschusses von 550.000 USD von den National Institutes of Health, um effiziente Methoden zu entwickeln, die die automatisierte strukturelle Klassifizierung, die Entdeckung von Merkmalen und die Strukturaufklärung von Naturprodukten erleichtern und eine Infrastruktur aufbauen, die mit den Daten der Gemeinschaft interagiert.
Originalveröffentlichung
Chen Zhang, Yerlan Idelbayev, Nicholas Roberts, Yiwen Tao, Yashwanth Nannapaneni, Brendan M. Duggan, Jie Min, Eugene C. Lin, Erik C. Gerwick, Garrison W. Cottrell & William H. Gerwick; "Small Molecule Accurate Recognition Technology (SMART) to Enhance Natural Products Research"; Scientific Reports; 2017



















































