Aufschlussreiche Partikeltrennungen
Amplifizierung unnötig: Ultrasensitive DNA-Quantifizierung per Lichtstreuung
Spuren von Biomolekülen wie DNA lassen sich mit einem neuen „dynamischen“ Verfahren nachweisen, das auf der Detektion einzelner Assoziation- und Dissoziationsereignisse von Gold-Nanopartikeln beruht: Ist die gesuchte DNA-Sequenz in der Probe, kann sie zwei Nanopartikel reversibel verbinden. Dies lässt sich anhand der veränderten Lichtstreuung in Echtzeit verfolgen. Wie die Wissenschaftler in der Zeitschrift Angewandte Chemie berichten, differenziert die Methode echte Signale von unspezifischen Ereignissen und kann sogar Abweichungen einzelner Basen erkennen.
© Wiley-VCH
Biomoleküle in extrem geringen Konzentration nachzuweisen und zu quantifizieren, ist von großer Bedeutung, etwa für eine möglichst frühe und genaue Diagnose von Krankheiten, die Begleitung von Therapien gegen Krebs, für forensische Untersuchungen sowie für hochempfindliche Tests auf biologische Kampfstoffe. Methode Nummer eins ist noch immer die auf einer enzymatischen Vervielfältigung von DNA basierende Polymerase-Kettenreaktion (PCR). Nachteil sind falsch positive Signale, die durch winzigste Verunreinigungen entstehen können.
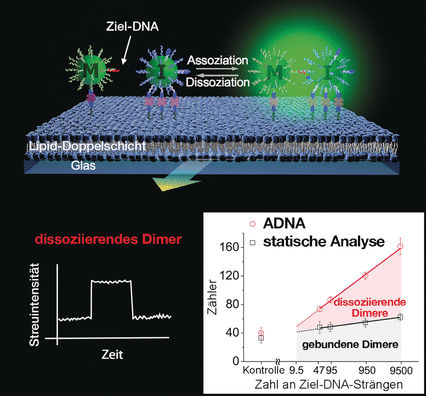
Wissenschaftler um Jwa-Min Nam von der Seoul National University (Südkorea) haben jetzt ein neues Konzept entwickelt, um extrem geringe Mengen an DNA nachzuweisen – ohne die Notwendigkeit einer Vervielfältigung oder Verstärkung des Signals und die Gefahr falsch positiver Ergebnisse. Es basiert auf der Detektion einzelner Bindungsereignisse. Da sich die Bindungspartner immer wieder trennen und neu zusammentun, potenziert sich die Zahl detektierbarer Ereignisse, während unspezifische Signale minimiert werden. Diese „assoziierende und dissoziierende Nanodimer-Analyse“, kurz ADNA, basiert auf einer Messung der Lichtstreuung von Gold-Nanopartikeln mit der Dunkelfeldmikroskopie.
Auf einen Glasträger wird eine Lipid-Doppelschicht aufgetragen, die Probe sowie zwei Sorten von Gold-Nanopartikeln aufgegeben. Eine Sorte trägt auf der Oberfläche Bindestellen, die sie auf der Lipidschicht fest verankern. Die andere Sorte bindet reversibel an die Lipidschicht und bleibt somit mobil. Beide tragen kurze einsträngige DNA-Stücke, die komplementär zu zwei verschiedenen Sequenzen der gesuchten DNA sind und diese daher binden können. Kommt nun ein mobiles Nanopartikel einem immobilisierten Nanopartikel sehr nahe, kann die Ziel-DNA beide Partikel zu einem Dimer verbrücken.
Sind zwei Nanopartikel auf diese Weise verbunden, koppeln auch ihre Schwingungen (Plasmonen). Dies verändert Intensität und Farbe des Streulichts und kann in Echtzeit detektiert werden. Vor allem die dynamische Analyse von Dimeren, die während der Beobachtungszeit dissoziierenden, ist der Schlüssel zu einer klaren Differenzierung zwischen Vorhandensein und Abwesenheit der Ziel-DNA. So ist der zeitliche Verlauf der Dissoziierung bereits deutlich anders für eine perfekt passende DNA und eine Sequenz, bei der eine einzelne Base verändert ist.
Auch in Anwesenheit anderer DNA, z.B. in einer Probe von menschlichem Blutserum, ließ sich eine Ziel-DNA in ultra-niedrigen Konzentrationen selektiv detektieren und verlässlich quantifizieren. Unter den verwendeten Testbedingungen lag die Nachweisgrenze bei ca. 46 DNA-Kopien.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.