Nobelpreis für Medizin 2022 geht an Svante Pääbo: Was macht uns zu einzigartigen Menschen?
Nobelpreis für seine Entdeckungen über die Genome ausgestorbener Hominiden und die menschliche Evolution
Die Menschheit war schon immer von ihren Ursprüngen fasziniert. Woher kommen wir, und wie sind wir mit denen verwandt, die vor uns kamen? Was unterscheidet uns, den Homo sapiens, von anderen Hominiden?
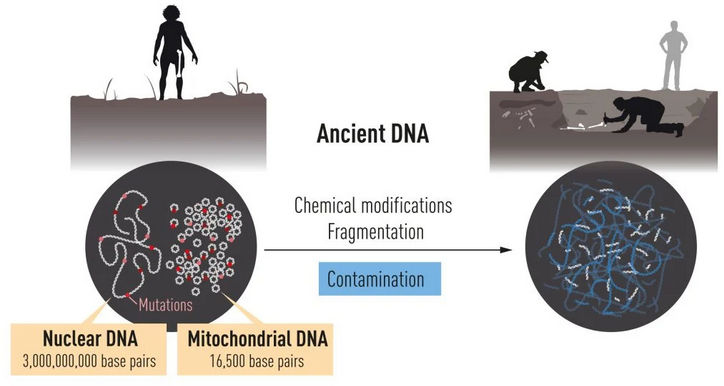
Die DNA ist in der Zelle in zwei verschiedenen Kompartimenten lokalisiert. Die Kern-DNA beherbergt den größten Teil der genetischen Information, während das viel kleinere mitochondriale Genom in Tausenden von Kopien vorhanden ist. Nach dem Tod wird die DNA im Laufe der Zeit abgebaut, so dass schließlich nur noch geringe Mengen übrig bleiben. Sie wird auch mit DNA von z. B. Bakterien und heutigen Menschen kontaminiert.
© The Nobel Committee for Physiology or Medicine. Illustrator: Mattias Karlén
Svante Pääbo hat mit seinen bahnbrechenden Forschungen etwas scheinbar Unmögliches geschafft: die Sequenzierung des Genoms des Neandertalers, eines ausgestorbenen Verwandten des heutigen Menschen. Außerdem machte er die sensationelle Entdeckung eines bisher unbekannten Hominiden, Denisova. Wichtig ist, dass Pääbo auch herausfand, dass ein Gentransfer von diesen nun ausgestorbenen Hominiden auf den Homo sapiens stattgefunden hat, nachdem sie vor etwa 70.000 Jahren aus Afrika ausgewandert waren. Dieser uralte Genfluss auf den heutigen Menschen ist heute physiologisch relevant, da er zum Beispiel die Reaktion unseres Immunsystems auf Infektionen beeinflusst.
Pääbos bahnbrechende Forschungen führten zur Entstehung einer völlig neuen wissenschaftlichen Disziplin: der Paläogenomik. Durch die Aufdeckung genetischer Unterschiede, die alle lebenden Menschen von ausgestorbenen Hominiden unterscheiden, bilden seine Entdeckungen die Grundlage für die Erforschung dessen, was uns zu einzigartigen Menschen macht.
Woher kommen wir?
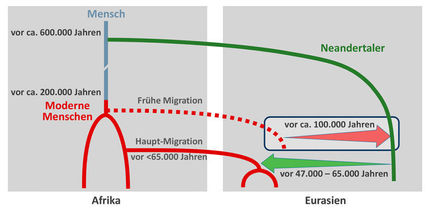
Die Frage nach unserem Ursprung und dem, was uns einzigartig macht, beschäftigt die Menschheit seit der Antike. Paläontologie und Archäologie sind wichtig für die Erforschung der menschlichen Evolution. Forschungen erbrachten den Nachweis, dass der anatomisch moderne Mensch, Homo sapiens , erstmals vor etwa 300.000 Jahren in Afrika auftauchte, während unsere nächsten bekannten Verwandten, die Neandertaler, sich außerhalb Afrikas entwickelten und Europa und Westasien von etwa 400.000 Jahren bis vor 30.000 Jahren bevölkerten, als sie ausstarben. Vor etwa 70.000 Jahren wanderten Gruppen von Homo sapiens von Afrika in den Nahen Osten ein und verbreiteten sich von dort aus in den Rest der Welt. Homo sapiens und Neandertaler lebten also in weiten Teilen Eurasiens über Zehntausende von Jahren nebeneinander. Doch was wissen wir über unsere Beziehung zu den ausgestorbenen Neandertalern? Anhaltspunkte könnten sich aus genomischen Informationen ergeben. Ende der 1990er Jahre war fast das gesamte menschliche Genom sequenziert worden. Dies war eine beachtliche Leistung, die spätere Studien über die genetischen Beziehungen zwischen verschiedenen menschlichen Populationen ermöglichte. Um jedoch die Verwandtschaft zwischen dem heutigen Menschen und dem ausgestorbenen Neandertaler zu untersuchen, wäre die Sequenzierung von genomischer DNA aus archaischen Proben erforderlich.
Eine scheinbar unlösbare Aufgabe
Schon früh in seiner Karriere war Svante Pääbo von der Möglichkeit fasziniert, die DNA der Neandertaler mit modernen genetischen Methoden zu untersuchen. Er erkannte jedoch bald die extremen technischen Herausforderungen, denn die DNA wird mit der Zeit chemisch verändert und zerfällt in kurze Fragmente. Nach Tausenden von Jahren sind nur noch Spuren von DNA übrig, und was übrig bleibt, ist massiv mit DNA von Bakterien und heutigen Menschen verunreinigt. Als Postdoktorand bei Allan Wilson, einem Pionier auf dem Gebiet der Evolutionsbiologie, begann Pääbo mit der Entwicklung von Methoden zur Untersuchung der DNA von Neandertalern, ein Unterfangen, das mehrere Jahrzehnte dauerte.
Im Jahr 1990 wurde Pääbo an die Universität München berufen, wo er als neu ernannter Professor seine Arbeit an archaischer DNA fortsetzte. Er beschloss, die DNA von Neandertaler-Mitochondrien zu analysieren - Organellen in Zellen, die ihre eigene DNA enthalten. Das mitochondriale Genom ist klein und enthält nur einen Bruchteil der genetischen Information in der Zelle, aber es ist in Tausenden von Kopien vorhanden, was die Erfolgsaussichten erhöht. Mit seinen verfeinerten Methoden gelang es Pääbo, einen Bereich der mitochondrialen DNA aus einem 40 000 Jahre alten Knochenstück zu sequenzieren. Damit hatten wir zum ersten Mal Zugang zu einer Sequenz von einem ausgestorbenen Verwandten. Vergleiche mit heutigen Menschen und Schimpansen zeigten, dass der Neandertaler genetisch anders war.
Die Sequenzierung des Neandertaler-Genoms
Da die Analysen des kleinen mitochondrialen Genoms nur begrenzte Informationen lieferten, nahm Pääbo nun die enorme Herausforderung an, das Kerngenom des Neandertalers zu sequenzieren. Zu dieser Zeit erhielt er das Angebot, ein Max-Planck-Institut in Leipzig, Deutschland, zu gründen. Am neuen Institut verbesserten Pääbo und sein Team kontinuierlich die Methoden zur Isolierung und Analyse von DNA aus archaischen Knochenresten. Das Forschungsteam nutzte neue technische Entwicklungen, die die Sequenzierung von DNA sehr effizient machten. Pääbo engagierte auch mehrere wichtige Mitarbeiter mit Fachwissen über Populationsgenetik und fortgeschrittene Sequenzanalysen. Seine Bemühungen waren erfolgreich. Pääbo schaffte das scheinbar Unmögliche und konnte 2010 die erste Genomsequenz des Neandertalers veröffentlichen. Vergleichende Analysen zeigten, dass der jüngste gemeinsame Vorfahre von Neandertaler und Homo sapiens vor etwa 800.000 Jahren lebte.
Pääbo und seine Mitarbeiter konnten nun die Beziehung zwischen Neandertalern und modernen Menschen aus verschiedenen Teilen der Welt untersuchen. Vergleichende Analysen zeigten, dass die DNA-Sequenzen von Neandertalern den Sequenzen von heutigen Menschen aus Europa oder Asien ähnlicher waren als denen von heutigen Menschen aus Afrika. Dies bedeutet, dass sich Neandertaler und Homo sapiens während ihrer jahrtausendelangen Koexistenz gekreuzt haben. Beim heutigen Menschen europäischer oder asiatischer Abstammung stammen etwa 1-4 % des Genoms von den Neandertalern ab.
Eine sensationelle Entdeckung: Denisova
Im Jahr 2008 wurde in der Denisova-Höhle im Süden Sibiriens ein 40 000 Jahre altes Fragment eines Fingerknochens entdeckt. Der Knochen enthielt außergewöhnlich gut erhaltene DNA, die das Team um Pääbo sequenzierte. Die Ergebnisse sorgten für Aufsehen: Die DNA-Sequenz war im Vergleich zu allen bekannten Sequenzen von Neandertalern und heutigen Menschen einzigartig. Pääbo hatte einen bisher unbekannten Hominiden entdeckt, der den Namen Denisova erhielt. Vergleiche mit Sequenzen von heutigen Menschen aus verschiedenen Teilen der Welt zeigten, dass es auch zwischen Denisova und Homo sapiens zu einem Genfluss gekommen war. Diese Verwandtschaft wurde erstmals in Populationen in Melanesien und anderen Teilen Südostasiens festgestellt, wo Individuen bis zu 6% Denisova-DNA tragen.
Die Entdeckungen von Pääbo haben zu einem neuen Verständnis unserer Evolutionsgeschichte geführt. Zu der Zeit, als der Homo sapiens aus Afrika auswanderte, lebten mindestens zwei ausgestorbene Hominidenpopulationen in Eurasien. Die Neandertaler lebten im Westen Eurasiens, während die Denisovaner die östlichen Teile des Kontinents bevölkerten. Während der Expansion des Homo sapiens außerhalb Afrikas und seiner Wanderung nach Osten traf er nicht nur auf Neandertaler, sondern auch auf Denisovaner und kreuzte sich mit ihnen.
Paläogenomik und ihre Bedeutung
Durch seine bahnbrechenden Forschungen begründete Svante Pääbo eine völlig neue wissenschaftliche Disziplin, die Paläogenomik. Nach den ersten Entdeckungen hat seine Gruppe die Analyse mehrerer weiterer Genomsequenzen von ausgestorbenen Hominiden abgeschlossen. Pääbos Entdeckungen haben eine einzigartige Ressource geschaffen, die von der wissenschaftlichen Gemeinschaft intensiv genutzt wird, um die menschliche Evolution und Migration besser zu verstehen. Neue leistungsfähige Methoden zur Sequenzanalyse deuten darauf hin, dass sich auch archaische Hominiden in Afrika mit Homo sapiens vermischt haben könnten. Bisher wurden jedoch noch keine Genome von ausgestorbenen Hominiden in Afrika sequenziert, da sich der Abbau archaischer DNA in tropischem Klima beschleunigt.
Dank der Entdeckungen von Svante Pääbo wissen wir jetzt, dass archaische Gensequenzen unserer ausgestorbenen Verwandten die Physiologie des heutigen Menschen beeinflussen. Ein Beispiel dafür ist die denisovanische Version des Gens EPAS1, die einen Vorteil für das Überleben in großen Höhen bietet und bei den heutigen Tibetern verbreitet ist. Andere Beispiele sind Neandertaler-Gene, die unsere Immunreaktion auf verschiedene Arten von Infektionen beeinflussen.
Was macht uns zu einzigartigen Menschen?
Der Homo sapiens zeichnet sich durch seine einzigartige Fähigkeit aus, komplexe Kulturen, fortschrittliche Innovationen und figurative Kunst zu schaffen, sowie durch die Fähigkeit, offene Gewässer zu überqueren und sich in alle Teile unseres Planeten auszubreiten. Neandertaler lebten ebenfalls in Gruppen und hatten große Gehirne. Sie benutzten auch Werkzeuge, die sich jedoch im Laufe von Hunderttausenden von Jahren nur wenig weiterentwickelten. Die genetischen Unterschiede zwischen dem Homo sapiens und unseren nächsten ausgestorbenen Verwandten waren unbekannt, bis sie durch Pääbos bahnbrechende Arbeit identifiziert wurden. Die laufende intensive Forschung konzentriert sich auf die Analyse der funktionellen Auswirkungen dieser Unterschiede mit dem Ziel, zu erklären, was uns zu einzigartigen Menschen macht.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.