Molekulare Waage auf biologischen Membranen: Mass-Sensitive Particle Tracking
Neue Methode bestimmt Orts- und Größenänderungen von unmarkierten Proteinen auf Membranen über ihre Masse
Ein Großteil biologisch relevanter Prozesse findet an Membranen statt. Die Dynamik dieser Prozesse in Echtzeit und ohne Störung des biologischen Systems zu studieren, ist bis heute eine große methodische Herausforderung. Ein Team um Petra Schwille, Direktorin am Max-Planck-Institut für Biochemie (MPIB), und Nikolas Hundt von der Ludwig-Maximilians-Universität München (LMU) hat hierfür nun eine neue Methode entwickelt: Mass-Sensitive Particle Tracking (MSPT). Mit MSPT lassen sich die Bewegungen und Reaktionen einzelner unmarkierter Proteine auf biologischen Membranen allein über ihre Masse bestimmen. Die Methode wurde in Nature Methods veröffentlicht.
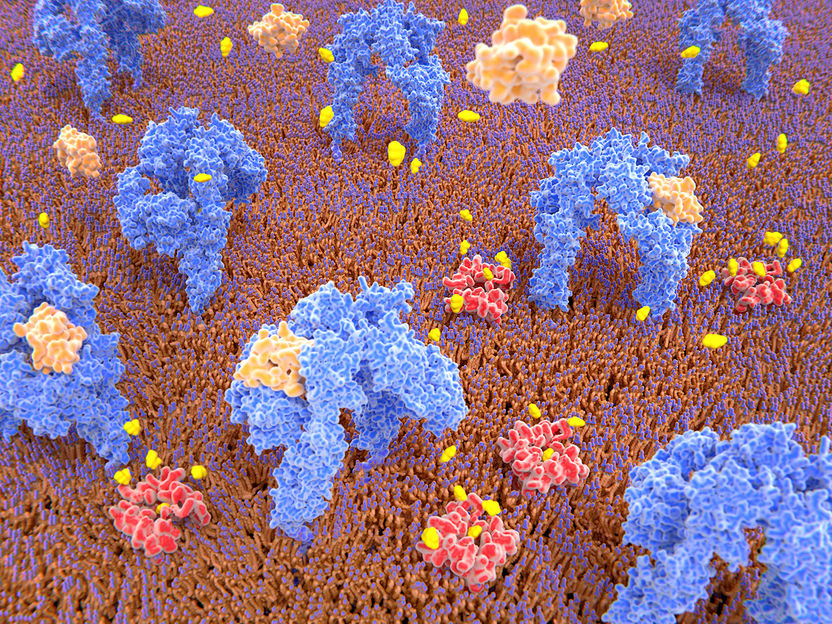
Mithilfe der neuen Methode Mass-Sensitive Particle Tracking (MSPT) können molekulare Masse, Bewegung und die Interaktion von Proteinen auf biologischen Membranen untersucht werden.
Juan Gärtner, AdobeStock
Zelluläre Prozesse an Membranen sind oft schnell und kurzlebig. Moleküle verbinden sich kurzzeitig, trennen sich wieder, interagieren mit unterschiedlichen Partnern und bewegen sich entlang der Membran oder durch sie hindurch. Daher ist es bei diesen Vorgängen wichtig, nicht nur statische Momentaufnahmen zu untersuchen, sondern die ablaufenden Prozesse in ihrer Dynamik zu verstehen. Wie jedoch gelingt dies methodisch? Petra Schwille vom MPIB und Nikolas Hundt von der LMU haben gemeinsam mit ihrem Team die Methode Mass-Sensitive Particle Tracking - MSPT entwickelt, die es erlaubt, Proteine während dynamischer Prozesse an Membranen zu analysieren.
Analyse dynamischer Prozesse an biologischen Membranen
Die Biophysiker gingen zunächst von neuen Entwicklungen in der Massenphotometrie aus, mit Hilfe derer bereits die molekulare Masse von unmarkierten Molekülen in Lösung bestimmt werden konnte. Neu bei MSPT ist, dass nun auch die Dynamiken von membranassoziierten Proteinen in ihrer biologisch plausiblen Umgebung nachverfolgt werden können. Dabei werden einzelne Proteine über ihre molekulare Masse identifiziert, ohne dass sie markiert werden müssen. Frederik Steiert, einer der Erstautoren der Publikation, sagt: „Wir können nun direkt an biologischen Membranen verfolgen, welche Masse einzelne Proteine haben, wie sie sich bewegen und wie sie interagieren. So können wir die Dynamik biologischer Systeme genauer untersuchen.“ Das Analysieren von dynamischen Vorgängen ist in der Biologie besonders wichtig, da viele Vorgänge an der Membran sehr kurzlebig sind.
Massenbestimmung durch Lichtstreuung
Auf welchen Prinzipien basiert die neue Methode? Wenn Licht auf ein Partikel trifft, wird dieses gestreut. Die Intensität des gestreuten Lichtes hängt von der Masse des Partikels ab. Am Mikroskop werden Videos aufgezeichnet, in denen einzelne Proteine auf Membranen direkt sichtbar gemacht werden. Mithilfe einer Analysesoftware können sie verfolgt und ihr Streusignal, und somit die Masse, bestimmt werden. Möglich ist dies aktuell bei Proteinen deren Molekulargewicht mindestens 50 kDa beträgt, also für einen großen Teil der bekannten Proteine. Ein weiterer Vorteil der neuen MSPT Methode besteht darin, dass Proteine nicht speziell gekennzeichnet - „gelabelt“ - werden müssen. Labeln lassen sich Moleküle beispielsweise durch das Anbringen fluoreszierender Markierungen. Allerdings besteht beim Labeln die Gefahr, dass Proteine in ihrer Funktion beeinträchtigt werden oder die fluoreszierenden Markierungen während der Untersuchung ausbleichen. Bei MSPT hingegen werden methodische Probleme, die durch das Labeln entstehen können, ausgeschlossen.
MinDE Proteinsystem
Um das Potenzial der Methode für biologische Fragestellungen zu demonstrieren, nutzten die Biophysiker ein etabliertes System aus dem Schwille-Labor: Das MinDE Proteinsystem aus dem Bakterium Escherichia coli (E. coli). Die Proteine MinD und MinE sind an der Zellteilung von E. coli beteiligt. Tamara Heermann, eine weitere Erstautorin, berichtet: „Die Methode ermöglicht es bisher nicht messbare Eigenschaften von dynamischen Systemen zu charakterisieren. Dadurch konnten wir nicht nur etablierte Kenntnisse über das Min-System verifizieren, sondern auch neue hinzugewinnen.“ Durch MSPT konnte das Team zeigen, dass die Komplexe aus MinD Proteinen größer sind als zunächst angenommen. Zudem liefern die Experimente erste Erkenntnisse, dass MinE als Verbindungsstück für MinD Proteine fungieren und so eine Membranfreisetzung von größeren Komplexen initiieren kann.
Wie im neuen Nature Methods Artikel berichtet wird, liefert MSPT wertvolle Erkenntnisse zur Aufklärung dynamischer Prozesse an biologischen Membranen. Die Forscher arbeiten jedoch kontinuierlich daran, die Methode noch weiter zu verbessern. So soll die Methode zukünftig auch auf integralen Membranproteinen anwendbar sein und die Detektion noch kleinerer Proteine ermöglichen.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Meistgelesene News
Weitere News von unseren anderen Portalen
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.

Themenwelt Proteinanalytik
Die Proteinanalytik ermöglicht einen tiefen Einblick in diese komplexen Makromoleküle, ihre Struktur, Funktion und Wechselwirkungen. Sie ist unerlässlich für die Entdeckung und Entwicklung von Biopharmazeutika, das Verständnis von Krankheitsmechanismen und die Identifizierung von therapeutischen Zielen. Durch Techniken wie Massenspektrometrie, Western Blot und Immunoassays können Forscher Proteine auf molekularer Ebene charakterisieren, ihre Konzentration bestimmen und mögliche Modifikationen identifizieren.




















































