3D-Modell menschlichen Lebergewebes ermöglicht bessere Diagnose
Geometrische und funktionelle Lebermodelle für optimierte Diagnose von nichtalkoholischer Fettlebererkrankung
Die nicht-alkoholische Fettlebererkrankung (NAFLD) ist in den Industrieländern eine der häufigsten chronischen Lebererkrankungen. Die histologische Analyse des Lebergewebes ist die einzige anerkannte Methode zur Diagnose und Abgrenzung verschiedener Stadien der Erkrankung. Die herkömmliche Histologie liefert jedoch nur zweidimensionale (2D) Bilder des Lebergewebes in niedriger Auflösung und vernachlässigt potenziell wichtige dreidimensionale (3D) strukturelle Veränderungen. Forscher des Max-Planck-Instituts für molekulare Zellbiologie und Genetik in Dresden (MPI-CBG) und des Universitätsklinikums Carl Gustav Carus Dresden (UKD) haben nun zusammen mit Kollegen der Technischen Universität Dresden (TUD) dreidimensionale geometrische und funktionelle Modelle des menschlichen Lebergewebes für verschiedene Stadien der NAFLD erstellt, die neue krankheitsbedingte Gewebeveränderungen sichtbar machen. Sie liefern neue Erkenntnisse über die NAFLD-Pathophysiologie und tragen zur hochauflösenden medizinischen Diagnose bei.
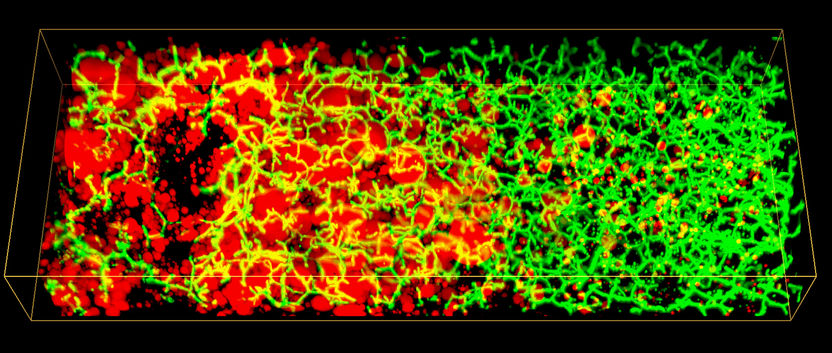
3D-Bild der Lipidtröpfchen (rot) und des Gallenkanalnetzwerks (grün) entlang der Leberläppchen eines Patienten mit nicht-alkoholischer Fettlebererkrangung (NAFLD).
Hernán Morales-Navarrete. Copyright: Segovia-Miranda et al. / MPI-CBG
Die nicht-alkoholische Fettlebererkrankung (NAFLD) ist durch die Anhäufung von Fett in der Leber in Verbindung mit einer Insulinresistenz gekennzeichnet, die nicht auf Alkoholkonsum zurückzuführen ist. NAFLD umfasst ein Spektrum von Lebererkrankungen, von einfacher Steatose („nicht-progressiv“ und reversibel) bis hin zu nicht-alkoholischer Steatohepatitis (NASH), die zu Zirrhose, Leberkrebs oder Leberversagen führen kann und letztendlich eine Transplantation erfordert. NAFLD ist die häufigste Ursache chronischer Lebererkrankungen. Im Jahr 2017 waren schätzungsweise 24 % der Weltbevölkerung von dieser Erkrankung betroffen. Die klassische histologische Analyse von Lebergewebe ist der Goldstandard zur Diagnose des Krankheitsverlaufes, hat aber mehrere Nachteile: Die niedrig aufgelösten 2D-Bilder des Lebergewebes ermöglichen nur eine semi-quantitative Auswertung, welche hängt von den Fähigkeiten des untersuchenden Arztes abhängt und damit sehr subjektiv sein kann. Vor allem liefern solche Bilder keine 3D-Informationen über Gewebestruktur und -funktion.
Um diese diagnostischen Einschränkungen zu überwinden, entwickelte das Forscherteam um MPI-CBG-Direktor Marino Zerial gemeinsam mit Kollegen aus der Medizinischen Klinik I, Fachbereich Gastroenterologie und Hepatologie am UKD, der Universitätskliniken Rostock und Kiel und dem Zentrum für Informationsdienste und Hochleistungsrechnen (ZIH) an der TUD dreidimensionale und räumlich aufgelöste geometrische und funktionelle Modelle des menschlichen Lebergewebes in verschiedenen Stadien der NAFLD. Die Leber hat eine komplexe dreidimensionale Gewebestruktur: Die Leber hat eine komplexe dreidimensionale Gewebestruktur und besteht aus funktionellen Einheiten, den Leberläppchen. Diese sind aus zwei ineinandergreifenden Netzwerken zusammengesetzt, den Sinusoiden für den Blutfluss sowie den Gallenkanälchen für die Gallensekretion und den Gallenfluss. Ein solcher Aufbau erschwert es, die dreidimensionale Organisation und die gesamte Gewebestruktur aus zweidimensionalen histologischen Bildern zu erkennen.
Im Jahr 2017 entwickelte die Forschungsgruppe von Marino Zerial ein Modell des Gallenkanalnetzwerks und des Gallenflusses der Mausleber durch eine umfassende 3D-Analyse der Gewebestruktur. Nun hat das Team die 3D-Organisation von menschlichem Lebergewebe untersucht. Obwohl in 2D-Bildern bereits einige Defekte zu erkennen sind, können die Veränderungen von Gallenkanälchen und Sinusoiden erst bei einer 3D-Rekonstruktion erkannt werden. Fabián Segovia-Miranda, der Erstautor der Studie, berichtet: „Neueste Fortschritte in der Multi-Photonenmikroskopie sowie verbesserte Methoden, um die Transparenz von Gewebe zu erhöhen, ermöglichten es dickere Gewebeschnitte hochauflösend abzubilden und damit 3D-Informationen zu gewinnen.“ Die digitale 3D-Rekonstruktion des Lebergewebes diente dann als Grundlage, um die Dynamik der Gallenflüssigkeit durch ein vom ZIH erstelltes Modell zu simulieren. Das erklärt Lutz Brusch von der Abteilung „Innovative Methoden des Computing“ am ZIH: „Während die Strömungsdynamik des Blutflusses durch die vergleichsweise großen Kapillaren bereits mittels Computersimulationen untersucht wurde, war dies für die Galle bisher unmöglich, da es an exakten geometrischen Daten des menschlichen Gewebes simultan über alle relevanten Skalen hinweg mangelte.“ Indem die Forscher Mikroskopie, digitale Bildrekonstruktion und Computermodellierung kombinierten, konnten sie eine Reihe von Zell- und Gewebeparametern identifizieren, die mit dem Fortschreiten der NALFD in Zusammenhang stehen. Fabián fügt hinzu: „Wir haben festgestellt, dass die Struktur des 3D-Gallenkanalnetzwerks im erkrankten Gewebe völlig anders ist. Solche strukturellen Veränderungen haben auch entscheidende funktionelle Auswirkungen. Durch eine personalisierte Simulation des Gallenflusses haben wir herausgefunden, dass der Gallenfluss in einigen kleinen Bereichen des Gewebes beeinträchtigt ist, was auch als Mikro-Cholestase bezeichnet wird.“
Marino Zerial, der auch dem Zentrum für Systembiologie Dresden (CSBD) angehört, gibt einen Ausblick: „Die High-Definition-Medizin ebnet den Weg für die Diagnose von Krankheiten wie NAFLD im Frühstadium, lange bevor Symptome auftreten. Sie hilft uns auch, molekulare krankheitsbedingte Mechanismen zu identifizieren, um neue Therapien zu entwickeln.“ Jochen Hampe, Professor am UKD, ergänzt: „Hier erlaubt uns die dreidimensionale Analyse des Lebergewebes völlig neue Einblicke in die Krankheitsmechanismen. So verstehen wir damit viel besser, wie beispielsweise der Gallenfluss und das Fortschreiten der Erkrankung zusammenhängen. Hier ergeben sich auch neue Ansätze für Therapien.“
Originalveröffentlichung
Fabián Segovia-Miranda, Hernán Morales-Navarrete, Michael Kücken, Vincent Moser, Sarah Seifert, Urska Repnik, Fabian Rost, Mario Brosch, Alexander Hendricks, Sebastian Hinz, Christoph Röcken, Dieter Lütjohann, Yannis Kalaidzidis, Clemens Schafmayer, Lutz Brusch, Jochen Hampeand Marino Zerial; "3D spatially-resolved geometrical and functional models of human liver tissue reveal new aspects of NAFLD progression"; Nature Medicine; 02. December 2019.



















































