Neues Verfahren zum Sortieren von Bakterien
Die Guten ins Töpfchen, die Schlechten ins Kröpfchen
Messungen, einen Kilometer tief im Meer. Forschung an Mammutbäumen, 100 Meter in der Höhe. Wissenschaftler haben es mitunter mit beachtlichen Dimensionen zu tun. Doch so groß muss es gar nicht immer sein: Mit klitzekleinen Bakterienkulturen beschäftigen sich Wissenschaftler am Leibniz-Institut für Naturstoff-Forschung und Infektionsbiologie – Hans-Knöll-Institut (HKI). Sie untersuchen Tröpfchen von 140 Pikoliter Volumen – das entspricht 0,00000014 Millilitern – und „durchleuchten“ sie in Rekordgeschwindigkeit.

Mit diesem Chip und den entsprechenden Computer-Algorithmen können HKI-Doktorand Emerson Zang und seine Kollegen pro Stunde 600.000 Bakterienkulturen untersuchen.
Tina Kunath
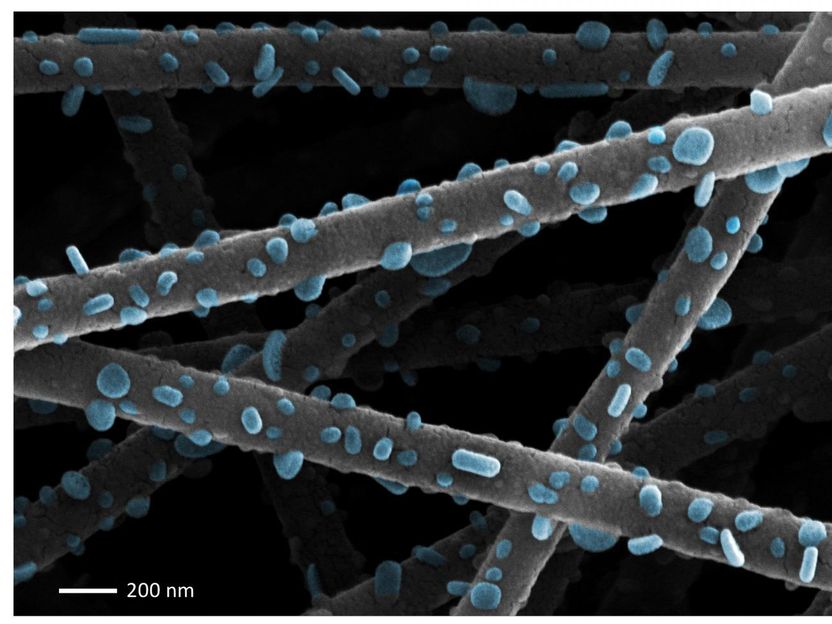
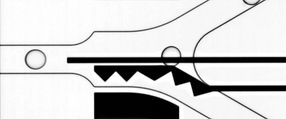
An dieser Stelle im Mikrofluidik-Chip werden mit Bakterien gefüllte Tropfen blitzschnell von leeren getrennt, pro Sekunde werden 100 solcher Fotos gemacht (50fach vergrößert)
Hans-Knöll-Institut

Es ist ganz normaler Erdboden, doch von ihm versprechen sich Wissenschaftler viel. Seit den 1940er Jahren erforschen sie Bodenproben auf der Suche nach neuen Bakterienarten, die Antibiotika produzieren. Ein jahrzehntelanges Erfolgskonzept. Bis heute konnten auf diese Weise viele Krankheiten zurückgedrängt werden. Doch immer mehr Erreger entwickeln Resistenzen gegen die verfügbaren Antibiotika.
Es sind also wieder Bodenproben gefragt. Doch so einfach wie vor 70 Jahren ist das längst nicht mehr. Der Erdboden enthält Unmengen an Bakterien. Neue Mikroben zu finden, die noch unbekannte Wirkstoffe produzieren, ist gar nicht so leicht, weiß Emerson Zang. Er fertigt am Biotechnikum des Hans-Knöll-Instituts seine Doktorarbeit an: „Die Pioniere von damals haben vor allem Bakterien entdeckt, die sich leicht kultivieren lassen. Wir wollen jetzt auch solche finden, die nicht so dominant sind, langsamer wachsen und bisher übersehen wurden. Antibiotika, die von diesen Bakterien produziert werden, sind für Krankheitserreger nämlich noch unbekannt.“ Eine Chance für die Entwicklung neuer Medikamente. Wissenschaftler des HKI und des benachbarten Instituts für Photonische Technologien (IPHT) entwickelten gemeinsam ein neues Verfahren, um solche versteckt lebenden Mikroben aufzuspüren.
Die Forscher nehmen Bodenproben und verdünnen die darin enthaltenen Bakterien in Wachstumsflüssigkeiten. Besonders interessieren sie sich dabei für sogenannte Actinobakterien, interessante Wirkstoffbildner, von denen bisher nur ein Prozent bekannt ist. Um herauszufinden, welche Bakterien in diesem Gemisch enthalten sind, wird die Flüssigkeit Tropfen für Tropfen in einen Sim-Karten-großen Chip geschleust – pro Tropfen eine Bakterienzelle mit dem Volumen von gerade einmal 140 Pikolitern. Durch ein Trennmittel bleiben die Tropfen abgekapselt voneinander, so dass sich die darin enthaltenen Mikroben nicht gegenseitig stören.
Im Anschluss wird den enthaltenen Bakterien Zeit gegeben sich zu vermehren. Um zu überprüfen, ob sie das getan haben, werden die Tröpfchen wieder durch den Chip geschleust und schließlich an einer Lichtschranke fotografiert. Die Geschwindigkeit, mit der dies geschieht, macht es für den menschlichen Beobachter unmöglich, die Ergebnisse selbst zu erfassen und weitere Entscheidungen zu treffen. Die Bioinformatikerin Susanne Brandes aus der Forschungsgruppe Angewandte Systembiologie untersucht die Fotos deshalb mit Hilfe von Computer-Algorithmen in Echtzeit: Haben sich die Bakterien vermehrt? Leere Tropfen werden blitzschnell aussortiert, Tropfen mit Bakterien bleiben im System und werden weiter für die Forschung genutzt.
Das Bemerkenswerte an dem Verfahren: Pro Sekunde können 100 Fotos gemacht werden. Das ergibt pro Stunde mehr als 600.000 untersuchte Kulturen. Beispiellos große Datensätze, die den entwickelten Prozess weltweit für Wissenschaft und Industrie interessant machen. Die Jenaer Wissenschaftler versuchen nun, mit dem Verfahren neue Bakterien zu finden, die bislang unbekannte Antibiotika produzieren – Hoffnung auf neue Medikamente.
In Kürze wird ihnen dazu ein neues Gerätesystem zur Verfügung stehen. Das Land Thüringen fördert die Entwicklung einer leistungsfähigen Forschungsinfrastruktur und bewilligte Prof. Erika Kothe vom Institut für Mikrobiologie der Friedrich-Schiller-Universität Jena knapp eine halbe Million Euro für das gemeinsam mit dem HKI bearbeitete Projekt „MikroInfra“. Es widmet sich unter anderem der weiteren Entwicklung dieser zukunftsträchtigen Technik.
Originalveröffentlichung
Zang E, Brandes S, Tovar M, Martin K, Mech F, Horbert P, Henkel T, Figge MT, Roth M (2013) Real-time image processing for label-free enrichment of Actinobacteria cultivated in picolitre droplets. Lab Chip 13(18): 3707-3713.