Virtueller Blick in das Zebrafischgehirn
Freiburger Forscher entwickeln Software zum automatischen Vergleich ganzer Gehirne
Es ist ein lange gehegter Traum des Menschen, das Gehirn als Ganzes zu begreifen und nicht nur die Funktion einzelner Nervenzellen zu verstehen. Diesem Ziel ist ein Team um Juniorprofessor Dr. Olaf Ronneberger vom Institut für Informatik der Universität Freiburg sowie Prof. Dr. Wolfgang Driever, Entwicklungsbiologe am Institut für Biologie I, nun ein Stück näher gekommen. Die Freiburger Forscher haben mikroskopische Aufnahmetechniken und eine Software entwickelt, mit denen sie in einem virtuellen dreidimensionalen Modell des Zebrafischgehirns sämtliche Gene und damit auch unterschiedliche auf die Nervenzellen einwirkende Faktoren anschauen und vergleichen können. Die Fachzeitschrift Nature Methods hat die Arbeit der Freiburger Forscher veröffentlicht.
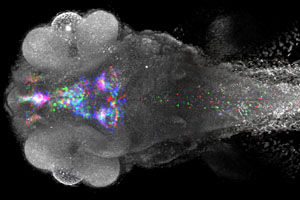
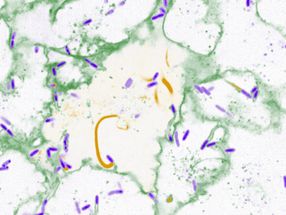
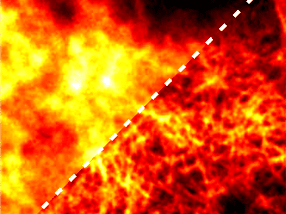
Das Bild zeigt die Genexpression (angefärbt) dreier verschiedener Larven eines Zebrafisches in einem virtuellen Standardgehirn.
Albert-Ludwigs-Universität Freiburg
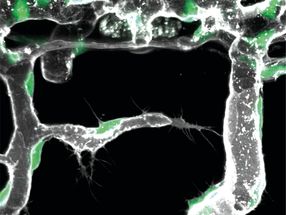
Ihr Virtual Brain Explorer für Zebrafische (ViBE-Z) ordnet im Experiment gewonnene genetische Daten automatisch bestimmten Punkten einer unter gleichen Bedingungen angefertigten Hirnaufnahme zu. Der Zebrafisch ist als Modellorganismus bei Biologen und Medizinern besonders beliebt, weil sich viele der an ihm gewonnenen entwicklungs- und neurobiologischen Erkenntnisse auf den Menschen übertragen lassen. Die sehr detaillierten Informationen der Hirnaufnahme werden verwendet, um die neuen Daten mit einem Standardgehirn in einer Datenbank zu vergleichen. Dies geschieht automatisch und mit einer Genauigkeit, die einzelne Zellen unterscheidet. Dazu entwickelte Olaf Ronneberger mit seinem Team neue Rechenverfahren, die sehr zuverlässig anatomische Strukturen im Zebrafischgehirn erkennen und in eine Datenbank des dreidimensionalen Gehirns übertragen.
Aus der Datenbank können die Wissenschaftler dann herauslesen, welche Gene in bestimmten Bereichen des Gehirns aktiv sind und wo welche Proteine die Hirnaktivität unter verschiedenen Versuchsbedingungen beeinflussen. So kann eine sehr große Anzahl von Faktoren, die gleichzeitig auf einen Hirnbereich einwirken, in integrierter Weise untersucht werden. „Damit verstehen wir das komplizierte Netzwerk Gehirn viel besser als in der Vergangenheit, und wir können erstmals unsere Daten mit denen aus einem Labor beispielsweise in Tokio oder Washington quantitativ im dreidimensionalen Raum vergleichen“, erklärt Wolfgang Driever. Die Freiburger Forscher werden ViBE-Z allen interessierten Kollegen zugänglich machen. Über ein Internetportal können Forscher dann überall auf der Welt ihre Bilder mitsamt ihrer genetischen Daten in das System einspeisen und nach etwa einer halben Stunde das daraus errechnete virtuelle Modell im Standardgehirn vom Freiburger Server herunterladen.
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Ab sofort nichts mehr verpassen: Unser Newsletter für Analytik und Labortechnik bringt Sie jeden Dienstag auf den neuesten Stand. Aktuelle Branchen-News, Produkt-Highlights und Innovationen - kompakt und verständlich in Ihrem Posteingang. Von uns recherchiert, damit Sie es nicht tun müssen.