Neue Methode erlaubt exakte Genkontrolle durch Licht
Werkzeugkasten der Forschung um ein neues Tool mit großem Potenzial ergänzt
Ein neuartiger optischer Schalter erlaubt es, die Lebenszeit genetischer „Abschriften“ exakt zu kontrollieren. Diese dienen der Zelle als Bauanleitung für die Herstellung von Proteinen. Die Methode wurde von Wissenschaftlern der Universitäten Bonn und Bayreuth entwickelt. Sie könnte die Untersuchung dynamischer Prozesse in lebenden Zellen deutlich vorantreiben.
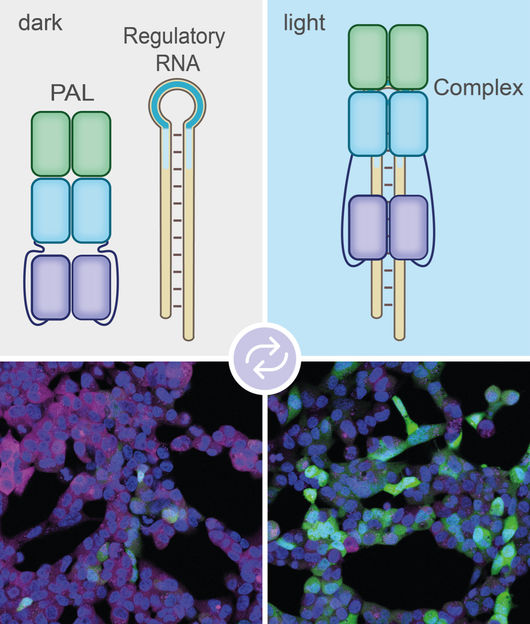
Bei Beleuchtung (rechts) bindet das PAL-Molekül an das Aptamer (blaue Schleife links oben). Das Label aus regulatorischer RNA kann daher nicht mehr an die mRNA binden. So wird diese nicht abgebaut.
© Sebastian Pilsl/AG Mayer/Uni Bonn
Bildlich gesprochen, enthält jede menschliche Zelle in ihrem Kern eine riesige Bibliothek mit Zehntausenden von Büchern, den Genen. Jedes dieser Bücher wiederum enthält die Bauanleitung eines Proteins. Wenn die Zelle ein bestimmtes Protein benötigt, wird von der entsprechenden Anleitung eine Abschrift hergestellt. Diese Kopien werden mRNAs genannt (RNA ist eine leicht abgewandelte Form der Erbsubstanz DNA).
Ein zellulärer Mechanismus sorgt dafür, dass die mRNA-Abschriften nach kurzer Zeit wieder „geschreddert“ werden. So ist sichergestellt, dass das Protein nur so lange produziert wird, wie es tatsächlich benötigt wird. Wissenschaftler sind schon vor einigen Jahrzehnten auf die Idee gekommen, diesen Schredder für eigene Zwecke zu nutzen: Indem sie bestimmten mRNAs ganz gezielt eine Markierung anheften, erreichen sie, dass die Abschriften gar nicht erst als Bauanleitung dienen, sondern direkt vernichtet werden – ein Prozess, der auch als RNA Silencing (RNA-Stummschaltung) bezeichnet wird. Der Zelle fehlt dann das entsprechende Protein. So lässt sich herausfinden, für welche Funktion es eigentlich zuständig wäre.
Bakterienmolekül als lichtabhängiger Schalter
Der Ansatz, den die Gruppen aus Bonn und Bayreuth nun publiziert haben, baut auf dieser Methode auf. Er ist allerdings längst nicht so grobschlächtig, sondern erlaubt eine weitaus differenziertere Kontrolle über die Lebensdauer der mRNA-Kopien. „Wir nutzen ein bakterielles Molekül, um das Zerschreddern der mRNA-Abschriften mit Hilfe von Licht zu steuern“, erklärt Prof. Dr. Günter Mayer, der am LIMES-Institut der Universität Bonn die Abteilung Chemische Biologie und Chemische Genetik leitet.
Das Bakterien-Molekül mit dem Kürzel PAL fungiert dabei als eine Art Schalter. Es ändert unter Einfluss von blauem Licht seine Gestalt. Dabei wird eine Tasche freigelegt, die an bestimmte Moleküle binden kann. „Wir haben eine riesige Bibliothek künstlich hergestellter kurzer RNA-Moleküle durchsucht, sogenannter Aptamere“, sagt Mayer. „Dabei sind wir auf ein Aptamer gestoßen, das sehr gut zu der Tasche im PAL-Molekül passt.“
Die Wissenschaftler haben dieses Aptamer nun an eine der molekularen Markierungen gekoppelt, die sich an mRNAs heften können und diese damit zum Abbau freigeben. „Wenn wir die Zelle mit blauem Licht bestrahlen, bindet PAL über das Aptamer an die Markierung und setzt sie damit außer Gefecht“, erläutert Mayers Mitarbeiter Sebastian Pilsl. „Die mRNA wird dann also nicht vernichtet, sondern in das entsprechende Protein übersetzt.“ Sobald die Forscher das blaue Licht ausschalten, lässt PAL die Markierung wieder los. Jetzt kann sie sich an die mRNA heften, die dann geschreddert wird.
Auf diese Weise können die Wissenschaftler künftig untersuchen, wo und wann ein Protein in einer Zelle genau benötigt wird – einfach, indem sie zu einem bestimmten Zeitpunkt einen Bereich der Zelle in blaues Licht tauchen und sich dann die Konsequenzen ansehen. In der aktuellen Studie haben sie das beispielsweise probeweise für Proteine umgesetzt, die bei der Regulation des Zellzyklus und der Zellteilung eine wichtige Rolle spielen. Die Verbindung aus Aptamer und Abbau-Markierung wird dabei auf gentechnischem Wege in die Zelle eingeschleust. Diese erzeugt das lichtabhängige Abbausignal danach also selbst; es muss nicht von außen zugeführt werden.
Gen-Abschriften lassen sich gezielt ausschalten
Das Aptamer lässt sich mit beliebigen Markierungen kombinieren, von denen jede wiederum als Schredder-Signal für eine bestimmte mRNA dient. „Mit dieser Methode lässt sich daher praktisch jedes mRNA-Molekül in der Zelle kontrolliert ausschalten“, betont Prof. Dr. Andreas Möglich von der Universität Bayreuth. In der jetzt veröffentlichten Pilotstudie funktionierte das Ganze ebenso einfach wie zuverlässig. Die Wissenschaftler sehen in ihrer Methode daher großes Potenzial für die Erforschung dynamischer Prozesse in lebenden Zellen und Organismen.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte
Zielgenaue Krebserkennung im Erbgut - Chemiker entwickeln Direktnachweis von Modifikationen an beliebigen Orten im Erbgut

Wenn Moleküle morsen
Kleinste Bausteine der Muskulatur verschwinden nach Schlaganfall - Erste Studie, die dieses Phänomen beim Menschen beobachtet
Vorstoß ins Unbekannte - Einsatz des Free Electron Lasers - Max-Planck-Physiker in Heidelberg starten Experimente mit ultrakurz gepulsten Laserstrahlen am "Free Electron Laser" (FEL) in Hamburg
Optische Distanzmessung mit Rekordgeschwindigkeit - Mit Solitonen-Frequenzkämmen aus optischen Mikrochips lassen sich 100 Millionen hochgenaue Distanzmessungen pro Sekunde durchführen

Langzeit-Supermikroskopie - Nanographene ermöglichen längere Beobachtungszeiten
Das Navi im Spermienschwanz: Proteine im Zick-Zack-Muster halten Spermien auf Kurs - Bewegung in 4D mikroskopisch untersucht
Schmallenberg-Virus erstmals sichtbar gemacht

Mini-Donut fängt Chlorid-Ionen - Formstabiler Makrocyclus als Chelatligand für Anionen

Effiziente Medikamentenentwicklung mit weniger Mäusen - Grosses Potential zur Reduktion der Versuchstierzahlen

Sensor in Größe eines Stickstoff-Atoms prüft Festplatten