Wirkstoffe gegen Krebs: Interaktive Software vereinfacht die Suche
Fraunhofer FIT präsentiert auf der analytica 2012 eine Software, die die Suche nach neuen Medikamenten gegen Krebs unterstützt. Ihre Architektur erlaubt eine parallele Bearbeitung mehrerer Analyseprozesse. Dadurch kann sie komplexe biologische Prozesse auch in großen Datenmengen besonders schnell verarbeiten und auswerten.
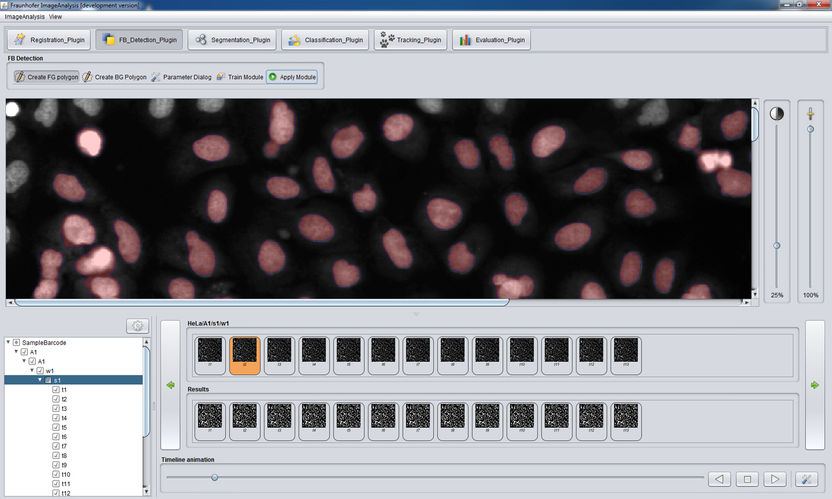
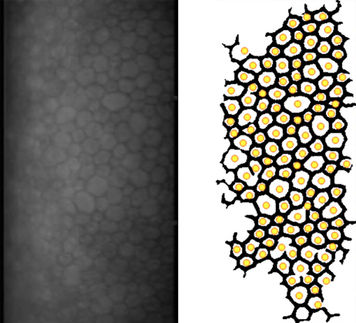
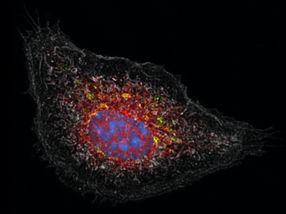
Aufgeräumte Oberfläche der Zeta Software: Mit wenigen Mausklicks wird die Software darauf trainiert, die Teilung von HeLa-Zellen zu erkennen.
© Fraunhofer FIT
Bei der Entwicklung neuer Wirkstoffe für die Krebstherapie setzen Forscher heute bildgebende Verfahren ein. Dabei entstehen große Mengen an Bilddaten. Ihre Auswertung ist zeitaufwändig und dauert oftmals länger als das Aufnehmen der Bilder. Hier hilft die Zeta Software, da sie einerseits die Auswertung vereinfacht und gleichzeitig auch große Datenmengen in kurzer Zeit verarbeiten kann. Mit wenigen Mausklicks wird die Software darauf trainiert, bestimmte Zellmuster zu erkennen und zu klassifizieren. Dadurch ist sie sehr flexibel für verschiedene biologische Fragestellungen einsetzbar.
Die Zeta Software des Fraunhofer FIT wurde jetzt speziell auf das so genannte Live-Cell-Imaging-Verfahren angepasst, das ermöglicht, Krebszellen über ihren gesamten Lebenszyklus zu beobachten und aufzuzeichnen. Bei diesem Verfahren werden Wirkstoffe auf lebende Zellen aufgebracht – und zwar viele Tausend Mal in verschiedenen Ansätzen. Wirkstoffe, die die Teilung von Krebszellen verhindern aber gleichzeitig die gesunden körpereigene Zellen unberührt lassen, haben das Potential zu einem Medikament. Diese Experimente laufen weitgehend automatisiert ab. Ein computergesteuertes Mikroskop erstellt Bildserien der lebenden Zellen. Anschließend verarbeitet eine robuste Bildanalyse die aufgenommenen Bilddaten. Hierbei unterstützt zukünftig die Zeta Software die Arbeit der Krebsforscher, denn sie ermöglicht, die Teilung von lebenden Krebszellen besser beobachten und quantifizieren zu können.
Eine besondere Herausforderung an die Bildanalyse bestand dabei darin, die einzelnen Phasen der Zellteilung zu differenzieren und sie miteinander in zeitlichen Bezug zu setzen. »Die Zellen müssen nicht nur als Objekte erkannt und den einzelnen Zellteilungsphasen zugeordnet werden, auch die Erkennung der zeitlichen Abfolge ist wichtig. Für jede Zelle wird also eine Historie angelegt«, so Prof. Dr. Thomas Berlage, Leiter des Forschungsbereichs Life Science Informatik am Fraunhofer-Institut für Angewandte Informationstechnik FIT.
Eine Besonderheit von Zeta ist seine Plug-In-Struktur, die die Software sehr flexibel adaptierbar macht. Vor Arbeitsbeginn werden über eine Konfigurationsdatei nur diejenigen Module in die Benutzeroberfläche geladen, die für die jeweilige Bildanalyse nötig sind. Mit dem Vordergrund-Hintergrund-Plug-In werden beispielsweise Zellobjekte vom Hintergrund getrennt. Hier bietet Zeta ein interaktives Verfahren an. Der Benutzer markiert mit der Maus einige Krebszellen und Hintergrundregionen. Die Software lernt anhand dieser Beispiele, die Zellen vom Hintergrund zu unterscheiden, und gibt direkt ein visuelles Feedback. Die Trainierbarkeit ist ein Vorteil der Software, weil dadurch auch neue Zelltypen erkannt werden, ohne dass Entwickler die Software anpassen müssen.
Weitere Plug-Ins ermöglichen etwa die Trennung von Zellclustern in einzelne Zellobjekte, können Zellen verschiedenen Zellzyklusphasen zuordnen oder eliminieren die bei Live-Cell-Imaging-Experimenten typischen Bewegungsartefakte.
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Analytik- und Labortechnik-Branche in Ihren Posteingang
Ab sofort nichts mehr verpassen: Unser Newsletter für Analytik und Labortechnik bringt Sie jeden Dienstag auf den neuesten Stand. Aktuelle Branchen-News, Produkt-Highlights und Innovationen - kompakt und verständlich in Ihrem Posteingang. Von uns recherchiert, damit Sie es nicht tun müssen.